Resistencia a Quinolonas
Las quinolonas de primera y segunda generación han sido muy utilizadas en clínica por sus buenas cualidades, las primeras hasta finales de los años 1970 y las de segunda generación durante las décadas de 1980 y 1990, llegándose a afirmar que estas últimas han sido probablemente los antimicrobianos más importantes en la quimioterapia antibacteriana durante dicho periodo (1)
Sin embargo, tienen una limitación importante: son poco activas frente a cocos grampositivos aerobios y apenas son eficaces sobre bacterias anaerobias. En consecuencia, no se han podido utilizar para el tratamiento de las infecciones por S. pneumoniae ni en infecciones por microorganismos anaerobios.
Conviene recordar brevemente que, para ejercer su efecto citotóxico, las quinolonas deben penetrar a través de la membrana bacteriana para alcanzar su diana celular, la DNA girasa o la topoisomerasa IV, y de esta forma producir la muerte celular. Por ello, en principio, las resistencias a las quinolonas pueden deberse a mutaciones que afecten cualquier paso de este proceso.
Primeramente, la DNA girasa es un complejo tetramérico, A2B2, formado por dos monómeros A (GyrA) y dos monómeros B (GyrB) codificados por los genes gyrA y gyrB. Las proteínas GyrA y GyrB son las dianas de las 4-quinolonas y de las cumarinas, respectivamente. Por otro lado, la topoisomerasa IV , otra enzima de tipo II, es también un tetrámero, C2E2, formado por dos subunidades C y dos E, codificadas por los genes parC y parE respectivamente. Para ejercer su acción, al igual que la DNA girasa, también precisa la presencia de las dos subunidades y de ATP. (2)
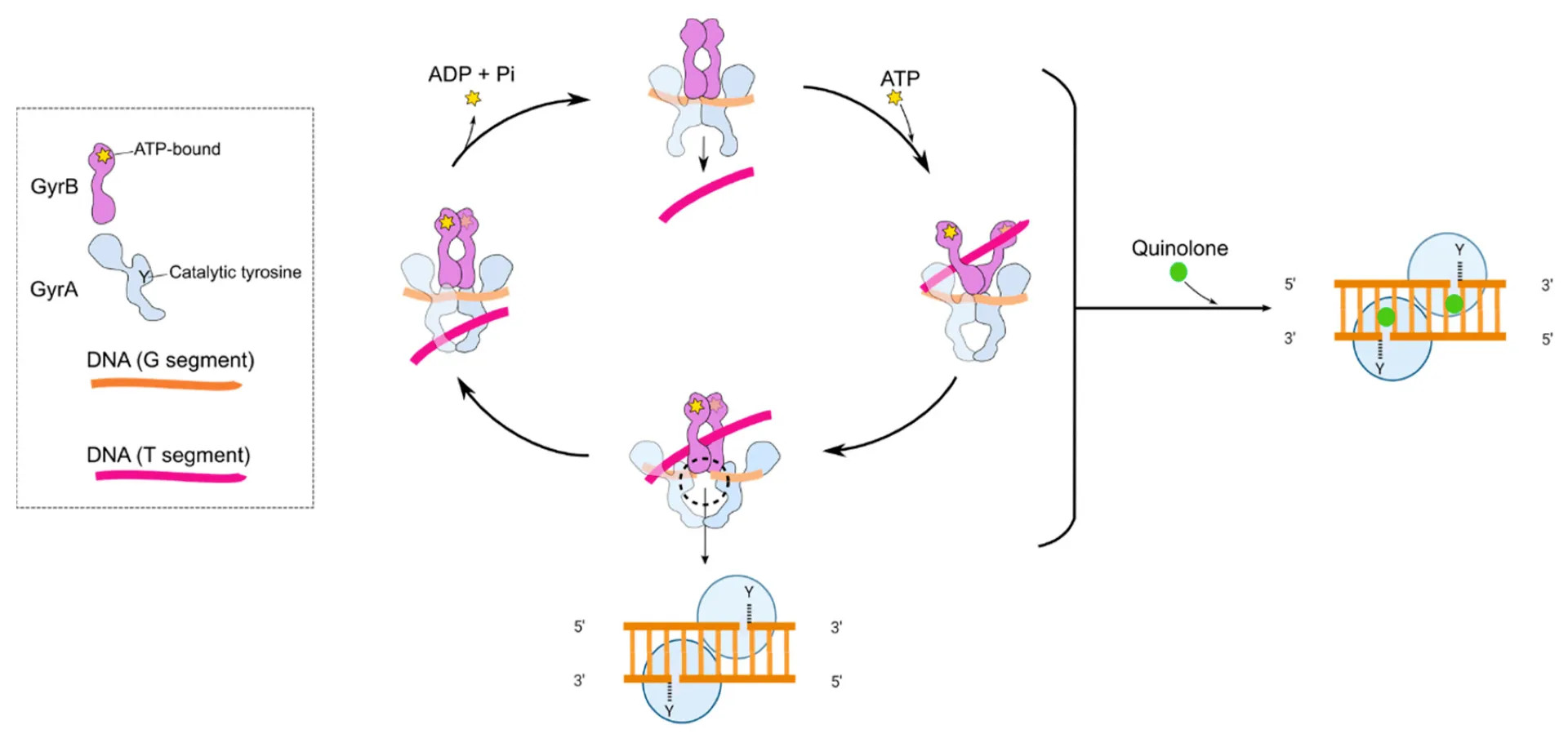
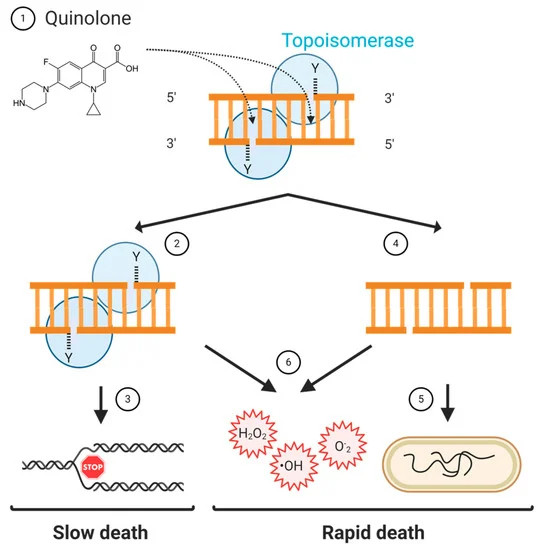
Figura 2. Modelo de letalidad de quinolonas. Las quinolonas estabilizan el complejo de escisión topoisomerasa-ADN en el que se produce una rotura de doble cadena. (2) Si el complejo de escisión no se resuelve, (3) la replicación y la transcripción no pueden ocurrir, lo que causa una muerte lenta de las células bacterianas. (4) Si se elimina la topoisomerasa, la rotura de la doble cadena queda libre y, si no se repara, (5) conduce a la fragmentación del cromosoma, lo que provoca una rápida muerte de las células bacterianas. (6) El complejo de escisión estabilizado, o la eliminación de la topoisomerasa del complejo de escisión, puede dar lugar a la acumulación de especies reactivas de oxígeno (ROS) que pueden causar una rápida muerte celular bacteriana.
Ahora bien, los aminoácidos codificados por los genes parC y parE son homólogos a los codifica dos por gyrA y gyrB, respectivamente. Secuencias similares se sitúan especialmente alrededor de la región de la DNA girasa conocida como región determinante en la aparición de resistencias bacterianas a las quinolonas (QRDR, del inglés quinolone resistance-determining regions) (42). Esta similitud en la secuencia de aminoácidos entre la DNA girasa y la topoisomerasa IV, especialmente en las posiciones productoras de resistencia a las quinolonas de la proteína GyrA de la girasa, implica que estos compuestos pueden inhibir la actividad de la topoisomerasa IV tanto como la de la DNA girasa
De tal forma, los mecanismos de resistencia a las quinolonas pueden agruparse en 2 categorías:
A. Resistencias de tipo cromosómica
Dan lugar a mutaciones de los cromosomas en segmentos definidos de los genes que se traducen en alteraciones en la girasa y la topoisomerasa IV, dando lugar a las QRDR (Regiones Determinantes de Resistencia a Quinolonas)
Los primeros estudios, demostraron que la resistencia a las quinolonas se produce normal mente por mutaciones en regiones definidas de las proteínas GyrA o GyrB. Las mutaciones en las regiones equivalentes de las proteínas ParC o ParE tienen lugar tras las producidas en la girasa y se asocian con altos grados de resistencia a estos fármacos. Sin embargo, a pesar de las quinolonas tienen como primera diana la DNA girasa en algunas bacterias grampositivas la situación es la inversa: las primeras resistencias tienen lugar por mutaciones producidas en la topoisomerasa IV, mientras que las mutaciones en la girasa proporcionan resistencias adicionales.
Los estudios posteriores se han realizado fundamental mente en S. pneumoniae, y se ha observado que en este microorganismo la actividad bactericida puede producirse a través de la girasa, la topoisomerasa IV o ambas, dependiendo de la estructura de la quinolona.
En función del orden de las consecutivas mutaciones QRDR en las topoisomerasas de mutantes resistentes seleccionados en S. pneumoniae, las quinolonas se pueden agrupar en tres clases:
- Grupo 1: Ciprofloxacino como principal exponente, levofloxacino, norfloxacino, pefloxacino y trovafloxacino. En este grupo, las mutaciones QRDR en la topoisomerasa IV se inducen antes que en la girasa, lo que sugiere que in vivo estos fármacos actúan preferentemente a través de la topoisomerasa IV.
- Grupo 2: Considera a Esparfloxacino, grepafloxacino, gatifloxacino, moxifloxacino y NSFQ-105 (homólogo del ciprofloxacino con un grupo 4-(4-aminofe nilsulfonil)-1-piperacinilo en C7), en donde se producen mutaciones en la girasa antes que en la topoisomerasa IV, lo que seña la como su diana principal a la girasa.
- Grupo 3: Clinafloxacino y el gemifloxacino, los cuales actúan a través de ambas dianas, girasa y topoisomerasa IV, pues aunque se generan en primer lugar mutaciones QRDR en gyrAo gyrB, esta mutación simple se produce con poca frecuencia y la resistencia es de bajo grado, lo que indicaría que ambas contribuyen de forma importante en la acción del fármaco. (3)
B. Resistencias basadas en la expulsión del fármaco.
Consiste en la expulsión del antibacteriano desde el medio intracelular al extracelular por acción de transportadores activos endógenos, impidiendo su acumulación, las quinolonas son sustratos de estos transportadores. Este sistema se sitúa en la membrana interna de los microorganismos y cataliza un proceso dependiente de energía ligado a un gradiente de protones.
Recientemente también se ha descrito un mecanismo de expulsión como causa de bajo grado de resistencia a las quinolonas en S. pneumoniae. A la bomba causante de esta acción se la denominó bomba PmrA.
La bomba PmrA (también conocida como pneumococcal multidrug resistance protein A) es un sistema de eflujo activo y pertenece a la familia de transportadores ABC (ATP-binding cassette), que están involucrados en el transporte de diversas moléculas a través de las membranas celulares. Su expresión es inducida por la presencia de quinolonas en concentraciones subinhibitorias, es decir, a niveles de estas sustancias que no son lo suficientemente altos como para tener un efecto antimicrobiano completo, pero que pueden afectar a las bacterias y contribuir a la resistencia o a la expresión de ciertos mecanismos, como ocurre en este caso.
De tal forma, este mecanismo de eflujo permite que Streptococcus pneumoniae elimine activamente los antibióticos, fuera de la célula. Al hacerlo, la bacteria puede reducir la concentración intracelular de estos compuestos y, en consecuencia, disminuir su efecto antimicrobiano. (3)
Referencias
(1) Blondeau, J.M. Expended activity and utility of the new fíuoro
quinolones: A review. Clin Ther 1999; 21: 3-40.
(2) Gellert, M., Mizuuchi, K., O'Dea, M.H., Nash, H.A. DNA gyrase: An enzyme that introduces superhelical turns into DNA. Proc Natl Acad Sci USA 1976; 73: 3872-3876.
(3) Taléns-Visconti R, Garrigues TM, Cantón y. E. Quinolonas y Streptococcus pneumoniae. Mecanismo de acción y resistencia [Internet]. Seq.es. [cited 2024 Apr 10]. Available from: https://seq.es/wp-content/uploads/2008/08/seq.es_seq_0214-3429_15_4_313.pdf
